En biologie, de nombreuses molécules d’ARN agissent comme des machines microscopiques sophistiquées. Parmi elles, les riboswitches fonctionnent comme de minuscules capteurs biologiques, modifiant leur forme 3D lorsqu’ils se lient à un métabolite spécifique. Ce changement de forme agit comme un interrupteur, souvent activant ou désactivant un gène en aval. La capacité à concevoir des interrupteurs artificiels en partant de zéro offrirait de grandes perspectives pour la biologie synthétique, la conception de médicaments et les nouveaux outils de diagnostic. Cependant, la conception d’une séquence capable de se replier de manière stable en deux formes différentes et de passer de l’une à l’autre est un défi extrêmement difficile à relever.
Une collaboration entre des chercheurs CNRS de l’ENS Paris, de l’Université Paris Cité, de l’École Polytechnique, et de l’Institut de Physique Théorique (IPhT), a permis de concevoir, grâce à une approche d’apprentissage automatique, des commutateurs ARN fonctionnels entièrement nouveaux. L’équipe a appliqué un modèle connu sous le nom de machines de Boltzmann restreintes (RBM) pour apprendre les « règles de conception » du domaine aptamère (1) d’une famille de riboswitches naturels. En entraînant les RBM sur des milliers de séquences naturelles, le modèle a appris à capturer les motifs complexes, les corrélations et les contacts secondaires et tertiaires (2) essentiels (comme les pseudoknots) que les modèles plus simples ne parviennent pas à détecter.
La RBM entraînée a ensuite été utilisée comme « générateur » pour créer 476 nouvelles séquences d’ARN, certaines différant jusqu’à 40 % des séquences naturelles connues. L’équipe a synthétisé et testé ces séquences artificielles à l’aide de sondes chimiques à haut débit (SHAPE et DMS). Les résultats ont été remarquables : environ un tiers des séquences conçues ayant obtenu les meilleurs scores ont fonctionné comme des commutateurs efficaces, modifiant leur conformation en réponse au métabolite cible (SAM), tout comme leurs homologues naturels.
Ce travail, qui vient d’être publié dans Nature Communications, représente une avancée significative dans la conception rationnelle des biomolécules allostériques. Il démontre comment les modèles génératifs, issus de la rencontre entre la physique statistique et l’IA, peuvent non seulement comprendre le langage complexe de la biologie, mais aussi l’utiliser pour écrire un nouveau « code » fonctionnel pour les molécules, ouvrant ainsi la voie à des outils moléculaires conçus sur mesure.
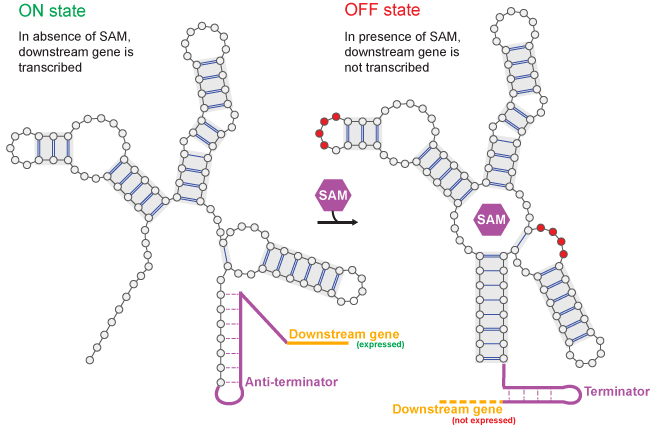
Un riboswitch agit comme un capteur biologique, changeant sa forme 3D d’un état « ON » (à gauche) à un état « OFF » (à droite) lorsqu’il se lie à un métabolite (SAM, hexagone violet). Les chercheurs du CNRS ont utilisé un modèle d’apprentissage automatique pour apprendre les règles de conception de ce commutateur, ce qui leur a permis de créer à partir de zéro des molécules artificielles fonctionnelles entièrement nouvelles.
1. Le domaine aptamère est la partie du riboswitch responsable de la reconnaissance et de la liaison d’un métabolite spécifique.
2. Les contacts tertiaires désignent des interactions particulières entre des sites éloignés le long de la séquence qui se retrouvent proches dans l’espace lorsque la molécule d’ARN est repliée.
En savoir plus :
– Article dans Nature Communications
– Article dans CNRS Physique
Informations complémentaires :
Laboratoire de physique de L’École normale supérieure (LPENS, ENS Paris/CNRS/Sorbonne Université/Université de Paris)
Auteur correspondant : Simona Cocco
Contact communication : L’équipe de communication